Startseite
Technologie
Entdecken Sie die Kraft der DNBSEQ™-Technologie
Eine Revolution in der DNA-Sequenzierung
Teilen

Der DNBSEQ™ Vorteil
Unsere DNBSEQ™ Technologie bietet mehrere wichtige Vorteile:
Der DNBSEQ™ Vorteil
Unsere DNBSEQ™ Technologie bietet mehrere wichtige Vorteile:
Erhöhte Genauigkeit: Die DNBSEQ™-Technologie kombiniert die geringe Fehlerakkumulation von DNA-Nanokugeln (DNB) mit der hohen Signaldichte von Array-Chips und führt zu einer deutlich verbesserten Detektionsgenauigkeit, insbesondere bei der Ganzgenomsequenzierung (WGS) und der Voll-Exomsequenzierung (WES).
Effiziente Vorbereitung und Verstärkung: Der DNBSEQ™-Prozess umfasst eine effiziente Einzelstrang-DNA-Zirkularisierung und DNB-Herstellung unter Verwendung der Rollenden-Kreis-Replikation (RCR). RCR reduziert Fehler, die bei der Amplifikation eingeführt werden, was zu einer erheblich verbesserten Sequenziergenauigkeit führt.
Optimierte Nutzung und hohe Durchsatzleistung: Das gemusterte Array und das DNB-Loading gewährleisten eine hohe Sequenziergenauigkeit, optimale Chip-Nutzung und effiziente Reagenzienverwendung. Dies führt zu hoher Durchsatzleistung und kostengünstiger Sequenzierung.
Fortgeschrittene cPAS-Technologie: Die im DNBSEQ™ integrierte Combinatorial Probe-Anchor Synthesis (cPAS)-Technologie ermöglicht eine schnelle und präzise Basenanrufung, wodurch die Reaktionszeit für die Sequenzierung erheblich verbessert wird.
Proprietärer Base-Calling-Algorithmus: MGIs proprietärer Subpixel-Registrierungsalgorithmus und der algorithmusbeschleunigte GPU ermöglichen die Extraktion von Bildintensität auf subpixeliger Ebene, was die Genauigkeit des Basenanrufs erheblich verbessert und die Datenverarbeitungsgeschwindigkeit dramatisch erhöht.
Wie DNBSEQ™ funktioniert
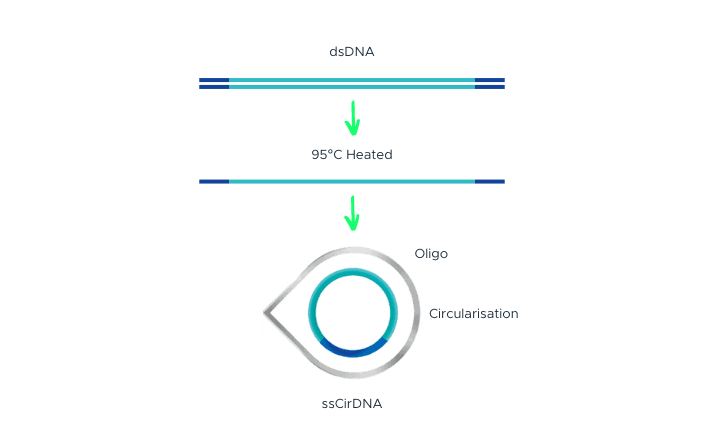
DNA Vorbereitung
Unser Prozess beginnt mit doppelsträngiger DNA, die wir erhitzen, um in einzelne Stränge zu trennen. Ein 'Splint-Oligonukleotid' bindet an diese Stränge und bildet einen 'nicked circle.' Dieser Kreis wird dann von DNA-Ligase versiegelt, um einen kontinuierlichen einsträngigen Kreis zu bilden. Diese präzise Methode gewährleistet zuverlässige Ergebnisse bei der DNA-Sequenzierung.
DNB Herstellung
DNA-Nanoballs (DNBs) werden durch Rolling Circle Replication (RCR) erzeugt, wodurch 300-500 Kopien aus einer einzelnen DNA-Vorlage entstehen. Diese Methode, die eine hochpräzise DNA-Polymerase verwendet, minimiert Replikationsfehler und verbessert die Genauigkeit unserer DNBSEQ-Sequenzierplattform.
DNB wird geladen
DNBs mit negativer Ladung haften dank ihres Phosphatrückgrats an der positiv geladenen Oberfläche der Durchflusszelle. Spezielle Puffer erhalten die Signalintegrität während des gesamten Sequenzierens, indem sichergestellt wird, dass in jedem Durchflusszellen-Spot nur ein DNB vorhanden ist, um die sequenzielle Effizienz zu optimieren.
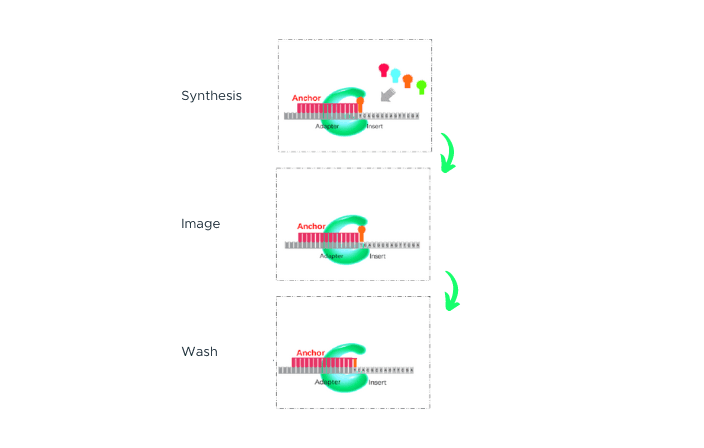
cPAS
In cPAS beginnt die Sequenzierung mit Primern, die sich an DNA-Nanokugeln (DNBs) binden, gefolgt von der Zugabe fluoreszierender dNTP-Sonden. Nach der Bildgebung und der Signalumwandlung in digitale Daten bereitet ein Regenerationsreagenz die DNBs für den nächsten Zyklus vor. Unser fortschrittlicher Sequenzierungspolymerase, ausgewählt aus Tausenden von Mutanten, beschleunigt die Sequenzierungsreaktion und verbessert Effizienz und Genauigkeit.
2. Strandvorbereitung
Nachdem der erste Strang sequenziert ist, synthetisieren wir den zweiten Strang unter Verwendung spezieller Primer und einer Polymerase mit Strangverdrängungsaktivität. Dieser Prozess erzeugt ein stärkeres Signal für den zweiten Strang und verbessert die Sequenzgenauigkeit durch optimierte Chemie.
Basisaufrufalgorithmus
Unser Basenanrufungsalgorithmus verwendet Signalintensität, um Basenanrufe und ihre Qualität zu bestimmen, und orientiert sich am PHRED-33-Standard. Durch einen proprietären Sub-Pixel-Registrierungsalgorithmus optimiert, erreicht er eine präzise Bestimmung von Basenanrufen auf subpixelgenauer Ebene und gewährleistet so branchenführende Geschwindigkeit und Genauigkeit bei der Datenverarbeitung